介绍 GeneCopoeia GeneHero™CRISPR-Cas9产品和服务为您的基因组编辑需求提供了完整、强大的解决方案。 产品和服务包括:
用于人、小鼠和大鼠的全基因组 sgRNA 克隆
HDR 供体克隆载体和定制的 HDR 供体克隆构建 Cas9稳定细胞系 Safe Harbor 基因敲入试剂盒及 ORF 敲入克隆 IndelCheck™ 插入缺失检测体系 sgRNA 文库 VividFISH™染色体FISH计数探针
优势
RNA导向的基因组DNA识别,无需考虑DNA甲基化
较ZFN和TALEN,有相同或更高的基因编辑效率
可同时编辑多个基因(多重靶向编辑)
设计简单快速,无需重复构建核酸内切酶
CRISPR工作原理 CRISPR-Cas系统(The clustered, regularly interspaced, short palindromic repeats-associated protein systems)是一个细菌及古细菌进化出来用以抵御病毒和质粒入侵的适应性机制。CRISPR-Cas系统的高效基因组编辑功能已被应用于多种生物,包括斑马鱼、小鼠、大鼠、秀丽隐杆线虫、植物及细菌。多个科研小组的研究都显示,与锌指核酸酶(ZFNs)和转录激活样效应核酸酶(Transcription activator-like effector nucleases, TALEN)相比较,CRISPR-Cas系统介导的基因组靶向实验在细胞或斑马鱼中具有相似甚至更高的效率。
CRISPR-Cas9体系的RNA-DNA识别机制为基因组工程研究提供了一项简便而强大的工具。该体系其中一个最重要的优势是Cas9蛋白可在多个不同的gRNA的引导下同时靶向多个基因组位点。
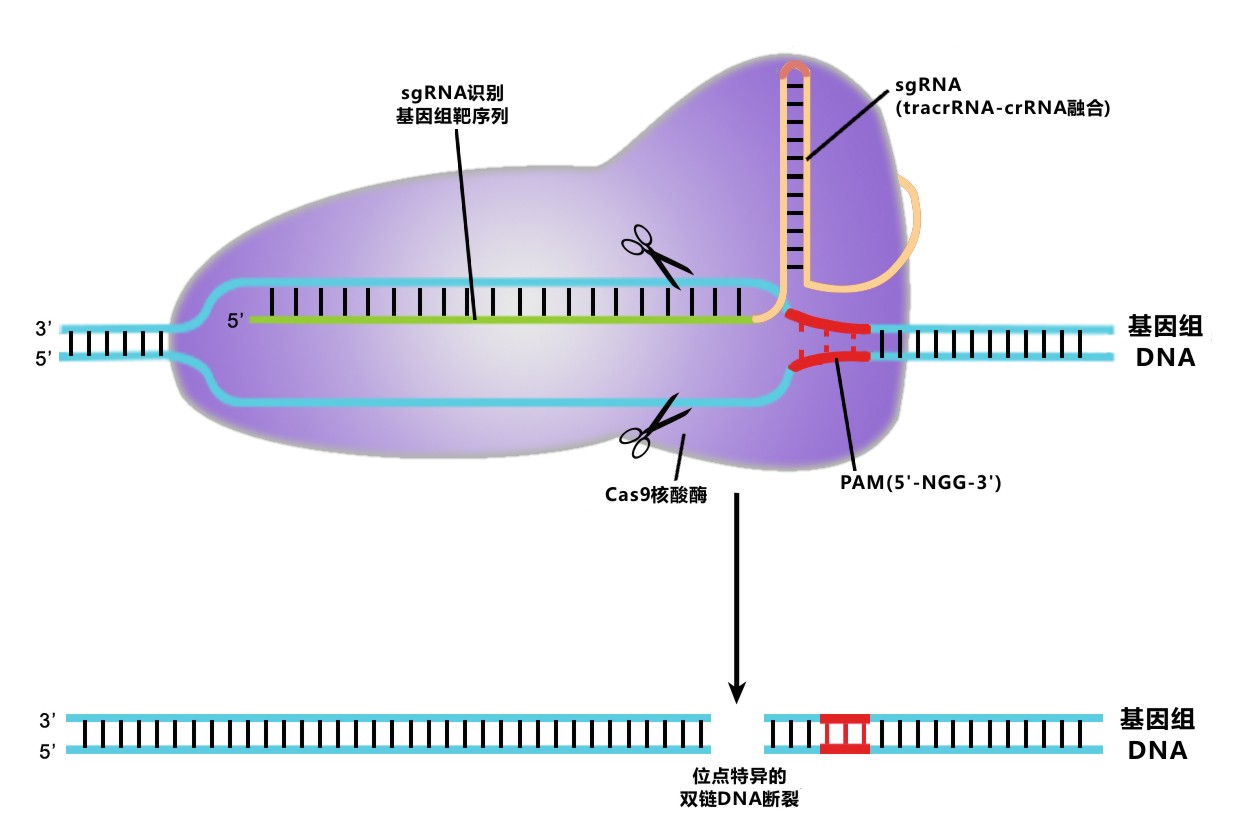
图 1. CRISPR/Cas9介导的基因组编辑原理图
在II型CRISPR系统中,CRISPR RNA(crRNA)与转录激活crRNA(Trans-activating crRNA, tracrRNA)退火形成的复合物能特异识别基因组序列,引导Cas9核酸内切酶在目的片段生成DNA双链断裂(double-strand breaks, DSBs)。 这个识别复合体可以通过融合crRNA与tracrRNA序列形成sgRNA(single-guided RNA)进行简化。基因组的靶序列中有长约20bp的片段与crRNA或sgRNA互补配对;靶序列末端的三核苷酸区域PAM(5’-NGG-3’)为Cas9识别位点,是实现剪切功能的关键。
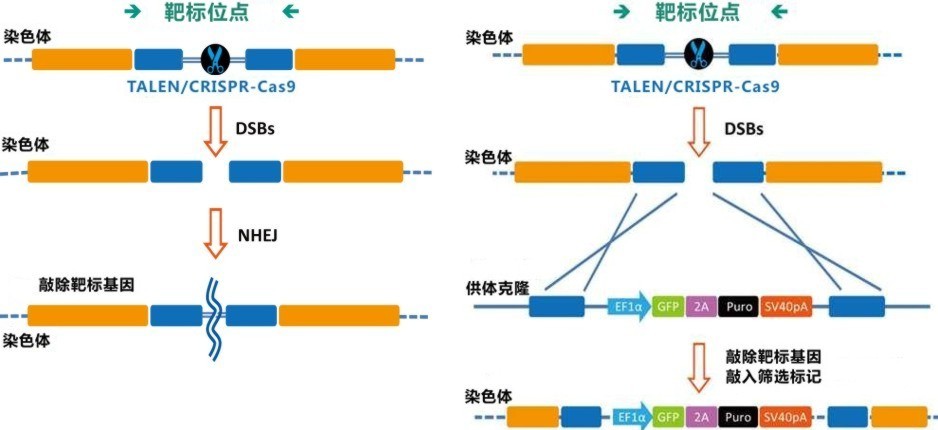
图2. CRISPR-Cas9介导的基因编辑。 (左):sgRNA引导Cas9核酸酶作用于基因组,形成的DSBs被非同源末端连接(NHEJ)机制修复;(右):sgRNA引导Cas9核酸酶作用于基因组,形成DSBs,同源重组(HR)作用下,供体质粒上的目标基因及筛选标记(或其他遗传元件)在断裂处被整合进基因组。
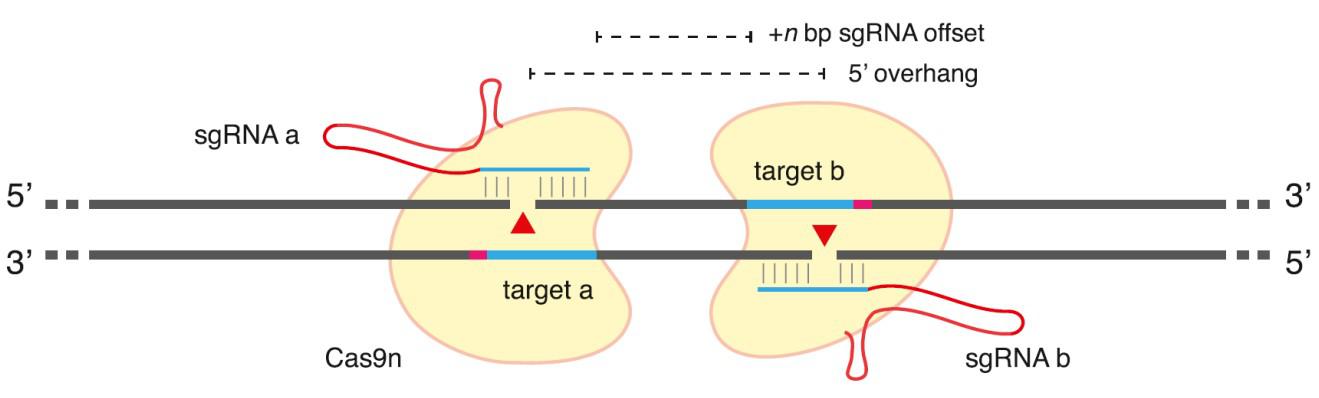
图3: 切口酶在结合链生产单链切口的原理图
TALEN 与 CRISPR-Cas9 体系对比
特性
TALEN
CRISPR-Cas9
识别方式
蛋白质 – DNA
RNA-DNA
甲基化敏感性
敏感
不敏感
染色质结构敏感性
敏感
敏感
脱靶效应
较少观察到脱靶效应
潜在脱靶效应高于 TALEN 及ZFN
多靶点操作
极少用
可用
基因组编辑应用 转录激活样效应因子 (TAL Effectors)和CRISPR-Cas9可高效且精确地改造细胞系及模式动物的基因组。具体的基因组编辑应用可见下表。
基因组修饰
描述
基因组编辑工具
供体DNA
内源基因标记
利用上融合标签(如荧光素酶、GFP等)跟踪内源性启动子活性或内源性蛋白的表达与定位。
TALEN 或CRISPR
必需
内源基因突变
在内源基因序列中引入点突变
TALEN 或CRISPR
必需
内源基因敲除
引入缺失或插入(如:筛选标记)敲除内源基因
TALEN 或CRISPR
若需建立敲除细胞系,则强烈建议使用。
内源基因表达激活
靶向激活内源基因表达
TALE-TF或 CRISPR-TF
不适用
内源基因表达抑制
靶向抑制内源基因表达
TALE-R 或CRISPR-R
不适用
Safe harbor 位点敲入 敲入外源ORF或其他遗传因子到人或小鼠基因组的safe harbor位点
TALEN或CRISPR
必需
参考文献
Horvath P, Barrangou R (January 2010). “CRISPR/Cas, the immune system of bacteria and archaea”. Science 327 (5962): 167–70.
Marraffini LA, Sontheimer EJ (February 2010). “CRISPR interference: RNA-directed adaptive immunity in bacteria and archaea” . Nat Rev Genet 11 (3): 181–190.
Hale CR, Zhao P, Olson S, et al. (November 2009). “RNA-Guided RNA Cleavage by a CRISPR RNA-Cas Protein Complex”. Cell 139 (5): 945–56.
van der Oost J, Brouns SJ (November 2009). “RNAi: prokaryotes get in on the act”. Cell 139 (5): 863–5. doi:10.1016/j.cell.2009.11.018.
Hale CR, Zhao P, Olson S, et al. (November 2009). “RNA-Guided RNA Cleavage by a CRISPR RNA-Cas Protein Complex” . Cell 139 (5): 945–56.
Jinek, M., Chylinski, K., Fonfara, I., Hauer, M., Doudna, J.A., and Charpentie E. (2012). A programmable dual-RNA-guided DNA endonuclease in adaptiv bacterial immunity. Science 337, 816–821.
Jiang, W., Bikard, D., Cox, D., Zhang, F., and Marraffini, L.A. (2013). RNA-guided editing of bacterial genomes using CRISPR-Cas systems. Nat.Biotechnol. 31, 233–239.
Hsu, P.D., Scott, D.A.,Weinstein, J.A., Ran, F.A., Konermann, S., Agarwala, V.,Li, Y., Fine, E.J., Wu, X., Shalem, O., et al. (2013). DNA targeting specificity of RNA-guided Cas9 nucleases. Nat. Biotechnol. Published online July 21, 2013.
Ran, et al. (2013). Double Nicking by RNA-Guided CRISPR Cas9 for Enhanced Genome Editing Specificity. Cell 154, 1380.
sgRNA克隆 GeneHero™ sgRNA设计与克隆定制服务 GeneCopoeia为客户感兴趣的基因提供sgRNA(single-guide RNA)设计与克隆定制服务。sgRNA克隆表达一个单链的融合sgRNA(由crRNA和tracrRNA融合而成)。当Cas9核酸酶存在时,sgRNA能识别靶标DNA序列并引导Cas9核酸酶剪切靶标位点,形成DNA双链断裂(DSBs),进而实现基因敲除、敲入及突变等基因组编辑。多个sgRNA克隆与一个Cas9克隆共转染可同时在基因组中编辑多个位点,使实验设计更高效、更灵活。
sgRNA载体类型
货号
载体
启动子
sgRNA
是否含有Cas9核酸酶基因
筛选标记/报告基因
载体图谱
价格(¥)
SG001
pCRISPR-SG01
U6
1 或多个
无,Cas9克隆单独出售*
Hygromycin
3960
SG002
pCRISPR-LvSG03
U6
1 或多个
无,Cas9克隆单独出售*
Puromycin / mCherry
3960
SG012
pCRISPR-CG02
U6
1
有,载体含有CBh启动子启动表达的Cas9核酸酶基因
N/A
3960
SG013
pCRISPR-CG04
U6
1或多个
有,载体含有CMV启动子启动表达的Cas9核酸酶基因
Neomycin / copGFP
3960
SG014
pCRISPR-CG07
U6
1
有,载体含有CBh启动子启动表达的Cas9核酸酶基因
copGFP
3960
SG015
pCRISPR-CG08
U6
1
有,载体含有CBh启动子启动表达的Cas9核酸酶基因
mCherry
3960
—
pCRISPR-CG12
U6
1或多个
有,载体含有CMV启动子启动表达的Cas9核酸酶基因
Neomycin / mCherry
—
—
pCRISPR-LvSG06
U6
1或多个
有,载体含有EF1a启动子启动表达的Cas9核酸酶基因
Puromycin
—
—
pCRISPR-LvSG09
U6
1或多个
有,载体含有EF1a启动子启动表达的Cas9核酸酶基因
Puromycin / eGFP
—
—
pCRISPR-LvSG10
U6
1或多个
有,载体含有EF1a启动子启动表达的Cas9核酸酶基因
Hygromycin
—
*配合单独表达Cas9 核酸酶 (Cat.No. CP-C9NU-01 , CP-LvC9NU-01 , CP-LvC9NU-02 , CP-LvC9NU-08 , CP-LvC9NU-09 , CP-LvC9NU-10 ) 或 Cas9 D10A 切口酶 (Cat.No.CP-C9NI-01 , CP-C9NI-02 ) 的 Cas9 克隆使用。
点击 获取单靶点或多靶点sgRNA设计和克隆服务的报价。
(A)
(B)
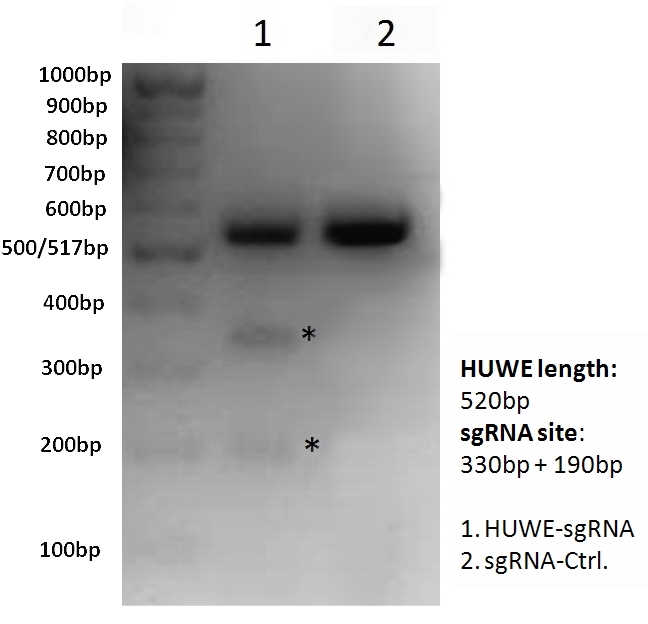
图4. HUWEI-sgRNA/Cas9靶向HEK293T细胞中的HUWEI基因。 (A)HUWEI-sgRNA与基因组PCR引物设计;(B)在6孔板中,HUWEI-sgRNA/Cas9克隆与sgRNA/Cas9对照克隆(泳道2)转染HEK293T细胞。转染40h后收集细胞,提取基因组DNA并用HUWE特异的PCR引物进行PCR扩增,凝胶纯化PCR产物,将纯化后的产物与缓冲液混匀,经变性、退火后在37℃水浴下T7 ENI剪切60min。HUWEI经PCR后的产物片段长度应为520bp,T7 ENI剪切后的产物应分别为330bp和190bp。
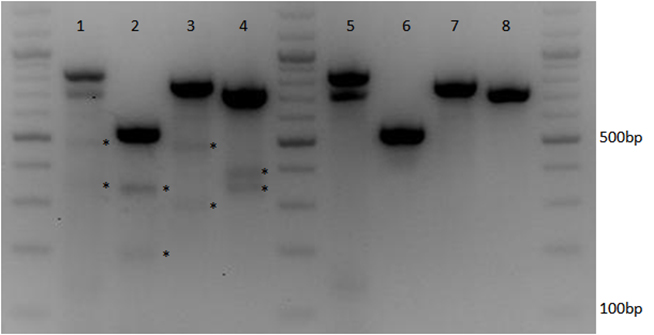
图5. CRISPR-Cas9多重靶向编辑多个基因。 实验组(1-4泳道)为Cas9表达克隆及分别表达靶向p53, HUWE1, NCL3 和 GFP基因的sgRNA克隆质粒共转染HEK293t GFP稳转细胞。对照组(5-8泳道)为Cas9表达克隆质及随机sgRNA表达克隆的质粒共转染HEK293t GFP稳转细胞。通过T7核酸内切酶检测分析基因组DNA各个靶点上同时存在的插入缺失。*号标记的条带显示Cas9核酸酶及分别靶向多个靶点的sgRNA都在目标靶点上有效地诱发了插入缺失突变(1-4道)。PCR产物条带及T7核酸内切酶酶切产物条带大小:GFP: 720bp (完整), 340bp + 380bp (酶切); NCL3: 765bp (完整), 295bp +470bp (酶切); HUWE: 520bp (完整), 190bp + 330bp (酶切); P53: 825bp (完整), 475bp + 350bp (酶切).
Cas9表达克隆 GeneHero™ Cas9 核酸酶及 D10A 切口酶表达克隆 Cas9核酸酶克隆表达按照人类密码子优化过的Cas9核酸酶基因。Cas9切口酶克隆表达一个Cas9 D10A 切口酶基因。该切口酶是一个 Cas9突变体,第10位氨基酸从野生型的天冬氨酸突变成了丙氨酸。
CRISPR-Cas9体系是由sgRNA和Cas9核酸酶构成的DNA靶向识别剪切复合体。sgRNA识别一段20bp、3端紧接着5′-N-G-G-3′ PAM位点的DNA靶序列,Cas9核酸酶在该靶序列上生成DNA双链断裂(DSB)。DNA双链断裂(DSB)会被细胞的非同源末端连接(NHEJ)或同源重组(HR)机制修复。NHEJ机制容易在修复的断裂位点引入插入/缺失突变,造成阅读框移码而产生基因敲除的效果。同源重组(HR)需要细胞内同时存在一段带有靶点同源序列的供体DNA作为修复模板,我们可以通过对供体DNA进行设计,在众多实验体系中实现基因敲除或者敲入突变修饰、报告基因等等一系列基因组修饰。
Cas9 D10A切口酶跟野生型的Cas9一样,也使用sgRNA识别靶序列。但 Cas9 D10A切口酶在靶序列上生成的是单链切口而非双链断裂。单链切口通常会由NHEJ和HR以外的机制完美地修复。但通过设计一对定向正确的sgRNA分别识别靶点两侧的DNA双链,我们可以诱导Cas9 D10A分别在靶点两侧的DNA双链上各生成一个单链切口,构成一个双链断裂(DSB),进而诱导NHEJ或HR修复机制。这种双切口策略常被用在要求低脱靶率的实验设计中。
GeneHero™ Cas9蛋白
User Manual
Technical Note
GeneHero™ Cas9 稳定表达细胞系 GeneCopoeia 提供 Cas9 核酸酶的稳定表达细胞系。这些细胞系能为您的 CRISPR 基因组编辑应用提供一个方便、高效的工具。
我们会提供预制的GeneHero™ Cas9 人类或小鼠稳定表达细胞系,如H1299或HEK293T。Cas9 核酸酶的基因已被稳定整合到这些细胞系基因组上的 safe harbor 位点。此外,我们也能将 Cas9 核酸酶基因稳定整合到您所指定的细胞系基因组。
更多产品信息,请访问我们的Cas9稳定表达细胞系 产品页面。
GeneCopoeia 也提供稳定细胞系构建服务。稳定细胞系构建服务请联系以下邮箱inquiry@igenebio.com ,或填写以下表格 。
资源 用户手册 GeneHero™ CRISPR-Cas9 用户手册
FAQs 基因组编辑常见问题及CRISPR FAQs
Protocols
技术说明 已发表文献
Pal, D., et al. (2023). H4K16ac activates the transcription of transposable elements and contributes to their cis-regulatory function. Nat Struct Mol Biol doi: 10.1038/s41594-023-01016-5 [GeneHero™ human AAVS1 safe harbor gene knockin kit ]
Naoki, I-T., et al. (2023). The Src-Family Kinases SRC and BLK Contribute to the CLDN6-Adhesion Signaling. Cells doi: 10.3390/cells12131696 [GeneHero™ sgRNA clones targeting mouse Blk; GeneHero™ sgRNA clones targeting mouse Scr ]
Talic, E-S., et al. (2023). RNA Methyltransferase METTL16’s Protein Domains Have Differential Functional Effects on Cell Processes. Curr Issues Mol Biol doi: 10.3390/cimb45070346 [GeneHero™ sgRNA control; GeneHero™ METTL16 Cas9-expressing clone ]
Wan, Y., et al. (2023). Nonmonotone invasion landscape by noise-aware control of metastasis activator levels. Nat Chem Biol doi: 10.1038/s41589-023-01344-z [GeneHero™ human AAVS1 safe harbor gene knock-in kits and clones ]
Schwarz, A.M., et al. (2023). Terpenes from Cannabis sativa Induce Antinociception in Mouse Chronic Neuropathic Pain via Activation of Spinal Cord Adenosine A2A Receptors. bioRxiv doi: 10.1101/2023.03.28.534594 [GeneHero™ Cas9-expressing clone ]
Li, C., et al. (2023). SUMO Proteomics Analyses Identify Protein Inhibitor of Activated STAT-Mediated Regulatory Networks Involved in Cell Cycle and Cell Proliferation. J Proteome Res doi: 10.1021/acs.jproteome.2c00557 [GeneHero™ PIAS1、PIAS2、PIAS3、PIAS4 sgRNA clones and negative control ]
Qin, Q., et al. (2023). CNTNAP4 signaling regulates osteosarcoma disease progression. NPJ Precis Oncol doi: 10.1038/s41698-022-00344-x [T7 Endonuclease I Assay Kit; SuperCut™ Nuclease ]
Lin, P., et al. (2023). TRAF6 regulates the abundance of RIPK1 and inhibits the RIPK1/RIPK3/MLKL necroptosis signaling pathway and affects the progression of colorectal cancer. Cell Death Dis doi: 10.1038/s41419-022-05524-y [GeneHero™ Cas9-expressing clone ]
Atobatele, A.G., et al. (2023). Canonical and truncated transglutaminase-2 regulate mucin-1 expression and androgen independency in prostate cancer cell lines. Cell Death Dis doi: 10.1038/s41419-023-05818-9 [GeneHero™ TGM2 sgRNA clones ]