Qihong Xu, Meng Zhang, Xueming Xu, and Ed Davis
简介
永生化的哺乳动物细胞系为生物医学和制药研究提供了方便的模型系统,但它们也经常携带3个甚至更多染色体拷贝或基因拷贝(Wistuba等,1998; Burdall等,2003;van Staveren等,2009)。例如,通常使用的人胚胎肾细胞系HEK293就是亚三倍体,染色体核型为64条。此外,HEK293和一些其它细胞系的倍性在细胞群中是不均等的。这为要求完全去除内源基因产物的CRISPR应用提出了特殊的挑战。因此,包括基因拷贝数鉴定在内的筛选方法细化对实验室培养的哺乳动物细胞基因组编辑是非常有益的。
荧光原位杂交(FISH)早已被用于非基因组编辑应用中的染色体和基因拷贝数鉴定,包括疾病诊断和预后(Landstrom&Tefferi,2006; Sarrate等,2010)。GeneCopoeia 近日发布的VividFISH™ 探针具有多种颜色(发射波长)和非常明亮的荧光。在本应用笔记中,我们演示了如何使用GeneCopoeia 的VividFISH™ 探针作为一个辅助技术,鉴定X染色体上携带5个有CRISPR基因敲除修饰的HDAC6基因拷贝的细胞系。我们的研究结果显示,将GeneCopoeia 的VividFISH™ 探针作为CRISPR基因组编辑工作实验流程的一部分,能极大地方便转染后的筛选工作。
结果
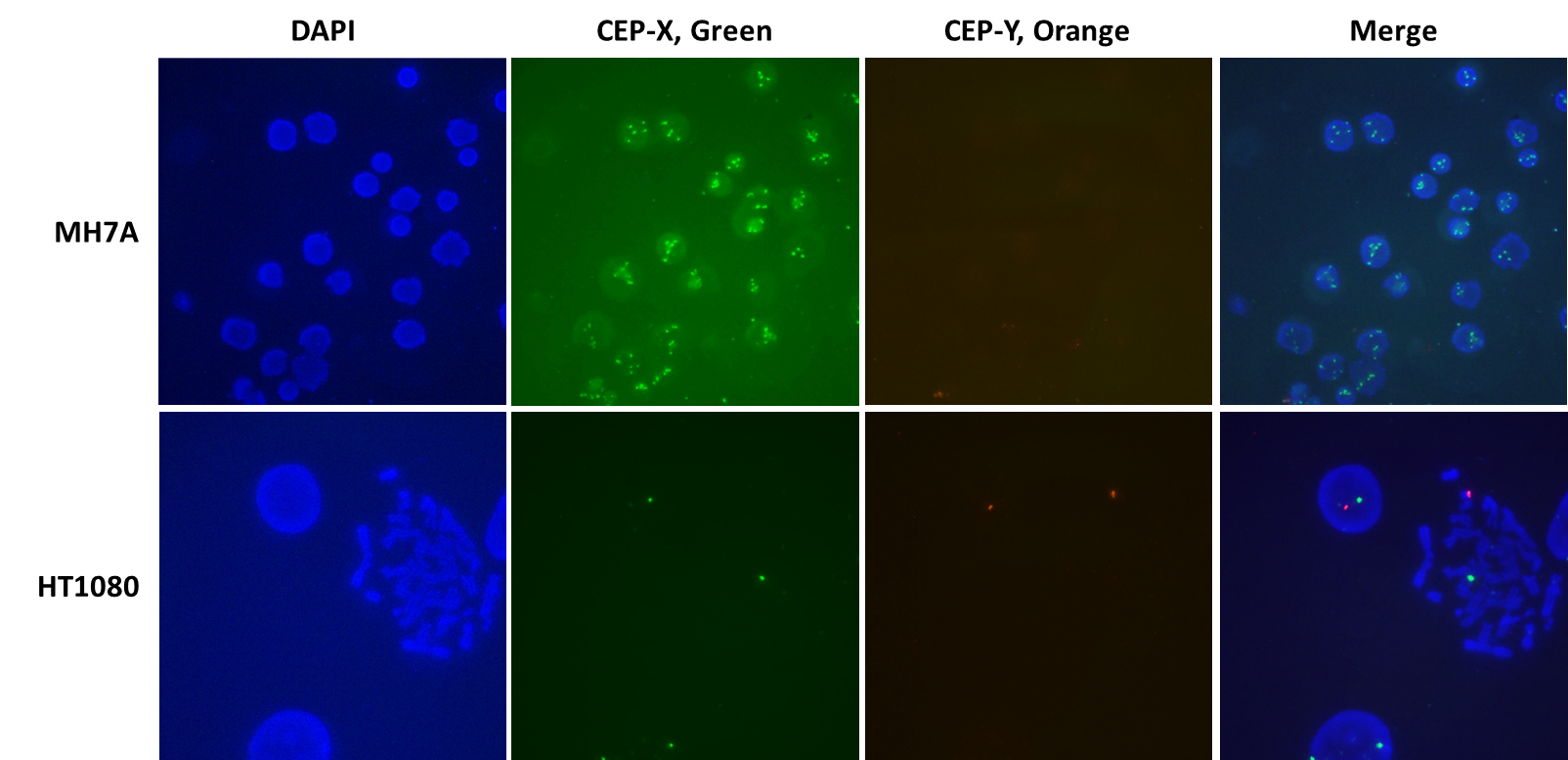
我们接手的这个实验项目要求完全敲除HDA6基因(NCBI geneID:10013)。该基因位于人滑膜成纤维细胞系MH7A的X染色体(Xp11.23)上。要注意某些细胞系会含有比正常的基因拷贝数(2)更多的基因拷贝,而我们也并不清楚MH7A细胞中的HDAC6基因有多少个拷贝,或者说,我们并不清楚在MH7A细胞中的X染色体拷贝数是多少。因此,我们决定使用X和Y染色体特异的VividFISH™ 着丝粒探针来鉴定在这些细胞中的X染色体拷贝数,以帮助我们筛选敲除了所有HDAC6 拷贝的MH7A稳定细胞系。通过在MH7A细胞上以及对照的二倍体雄性HT1080细胞系上使用VividFISH™ 探针,我们确定了大部分MH7A细胞显示5个X染色体荧光信号,无Y染色体特异性信号(图1)。而对照二倍体HT1080细胞仅显示一个X染色体信号和一个Y染色体信号(图1)。以上结果表明,MH7A细胞携了5个X染色体拷贝。
图1. 使用VividFISH™着丝粒探针鉴定MH7A细胞的X染色体拷贝数。未经基因组编辑的MH7A细胞(顶部)和二倍体HT1080细胞系(底部)被分别固定在显微镜玻片上并与VividFISH™ X染色体(绿色;GeneCopoeia 货号FP111)探针以及Y染色体(橙色;GeneCopoeia 货号FP117)探针杂交。只有雄性细胞系HT1080与Y染色体探针成功杂交。细胞核用DAPI(蓝色)染色。
在HDAC6基因敲除方面,我们用靶向该基因编码区ATG起始子附近区域的sgRNA表达质粒(同时表达Cas9核酸酶)转染转MH7A细胞。该质粒表达的sgRNA和Cas9核酸酶会在细胞基因组生产DNA双链断裂(DSB),介导插入或缺失(插入缺失)突变,以诱发阅读框移码。
转染后我们分离了MH7A单克隆稳定细胞系。为了减少需要进行插入缺失筛选的单克隆细胞系数目,我们先使用了GeneCopoeia IndelCheck™系列的T7核酸内切酶 I 试剂盒进行检测和筛选(数据未示出)。
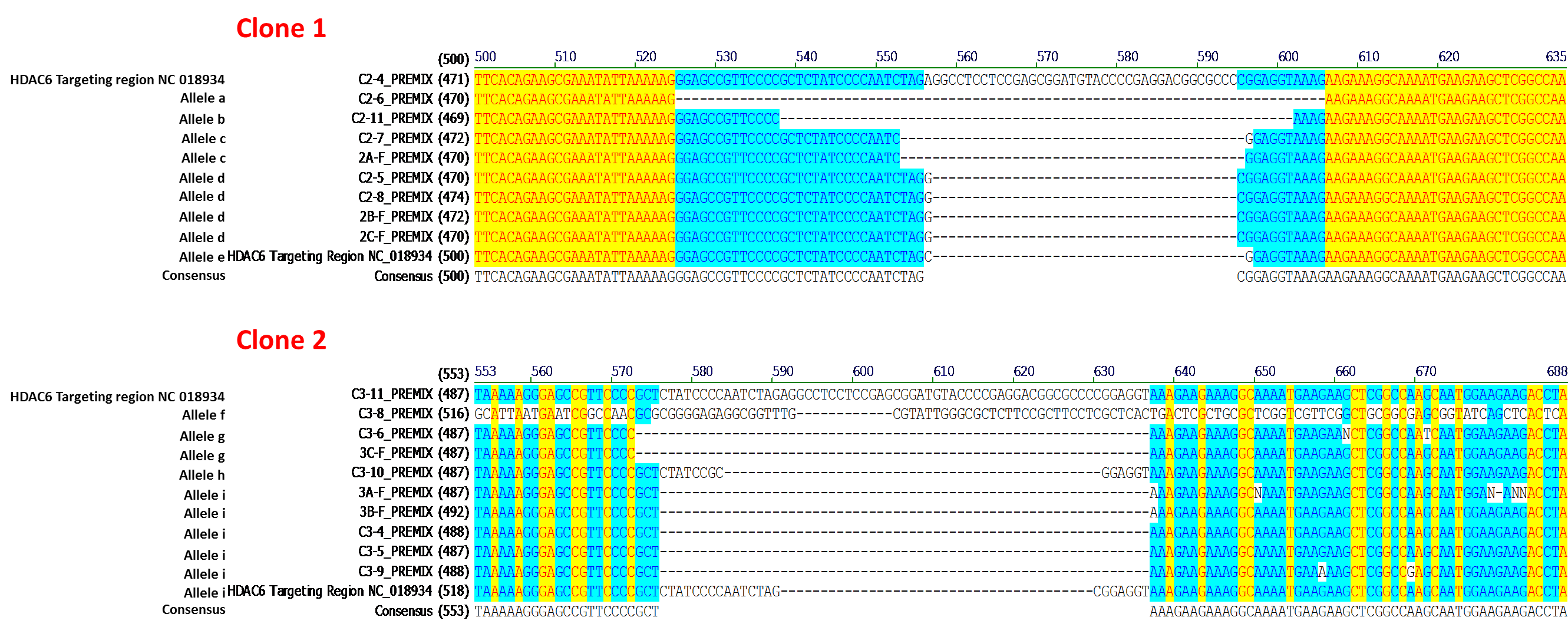
IndelCheck™ 的T7核酸内切酶I检测试剂盒使用错配酶切检测(邱等人,2004)方法。酶切检测需要先获得目标MH7A细胞系靶序列的PCR产物。这些带有插入缺失突变的PCR产物被亚克隆到E.coli中并送测。测序数据分析的结果显示,每个分离出来的MH7A单克隆细胞系都携带1到5个CRISPR基因组编辑介导的插入缺失突变。其中两个细胞系的测序结果显示它们各自带有5个不同的移码突变(图2)。这些结果表明,如图1所示,MH7A细胞携带至少5个HDAC6基因拷贝,与 VividFISH™探针鉴定结果一致。
图2. 在转染靶向 HDAC6(NCBI geneID:10013)的GeneCopoeia sgRNA 表达质粒后,分别分离两个MH7A 单克隆稳定细胞系,并进行测序分析。使用IndelCheck™ 检测体系(GeneCopoeia 货号:ICPE-050)取得的靶点PCR产物被用于构建E.coli 亚克隆并送测。每个MH7A来源细胞系都带有5个不同的移码突变,突变均由一次转染生成。
为了确定含有插入缺失突变的MH7A细胞系是否还携带HDAC6基因的拷贝(已被修饰或未被修饰的),我们从此前检测到带有5个不同插入缺失突变的3个MH7A细胞系上构建了更多亚克隆并送测。从这三个细胞系的亚克隆测序结果看,我们都没有检测到更多已被修饰或未经修饰的HDAC6等位基因(未示出数据)。以上结果表明,我们能够使用CRISPR在一个带有5个目的基因拷贝的细胞系实现同时敲除五个等位基因,而VividFISH™ 探针为我们筛选出这些基因敲除细胞系提供了宝贵的工具。
讨论
上述实验结果结果表明,GeneCopoeia 的 VividFISH™ 探针是一种功能强大、用途广泛的染色体计数工具,对在实验室培养的永生化哺乳动物细胞系进行的CRISPR基因组编辑实验有促进作用。
在使用着丝粒FISH探针来估算基因拷贝数时,有一个问题需要注意:探针可能检测不出基因倍增和基因易位,这会导致低估基因拷贝数。我们不建议用FISH着丝粒探针鉴定准确的基因拷贝数。我们建议的是您可以使用VividFISH™ 的检测结果对目标染色体拷贝数进行估算,以估算需要分离多少个细胞系单克隆用于筛选修饰阳性的细胞系。这些单克隆细胞系的测序结果可以与VividFISH™的检测结果结合使用,以最终确定实际的基因拷贝数。因此,我们推荐您在开始CRISPR基因组编辑细胞实验前,加入用VividFISH™探针鉴定染色体拷贝数的步骤。
参考文献
Burdall, et al. (2003). Breast cancer cell lines: friend or foe? Breast cancer research 5, 89.
Landstrom & Tefferi (2006). Fluorescent in situ hybridization in the diagnosis, prognosis, and treatment monitoring of chronic myeloid leukemia. Leukemia & Lymphoma 47, 397.
Qiu, et al. (2004). Mutation detection using Surveyor™ nuclease. Biotechniques 36, 702.
Sarrate, et al. (2010). Role of sperm fluorescent in situ hybridization studies in infertile patients: Indications, study approach, and clinical relevance. Fertility and Sterility 93, 1892.
van Staveren, et al. (2009). Human cancer cell lines: Experimental models for cancer cells in situ? For cancer stem cells? Biochimica et biophysica acta 1795, 92.
Wistuba, et al. (1998). Comparison of features of human breast cancer cell lines and their corresponding tumors. Clinical Cancer Research 4, 2931.
| Copyright ©2016 GeneCopoeia, Inc. www.genecopoeia.com TNGE8-020516 |